Questo articolo fa riferimento al laboratorio Modelli per le epidemie 2.0 realizzato da Alessio De Cesare, Stefano Manauzzi, Lorenzo Pazienza, Francesco Scaldarella, Lavinia Soldera. Il foglio di calcolo descritto nel testo si può raggiungere seguendo questo collegamento mentre il materiale a supporto è raccolto in un file zip.
Nel corso del laboratorio Modelli per le epidemie 2.0 (descritto nel fascicolo omonimo) abbiamo riscritto e migliorato SIR_ECovid, l’app creata nel 2020 e successivamente più volte aggiornata: la nuova versione si chiama SIR_ECHovid e nel nome si aggiunge la sezione della classe che ha lavorato quest’anno al progetto RiA, la 4°H.
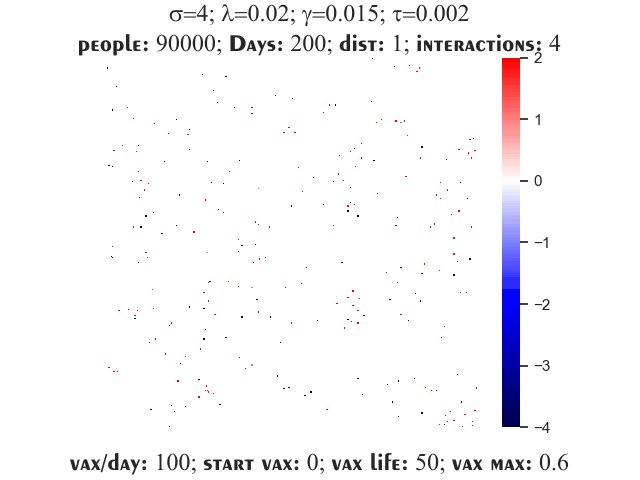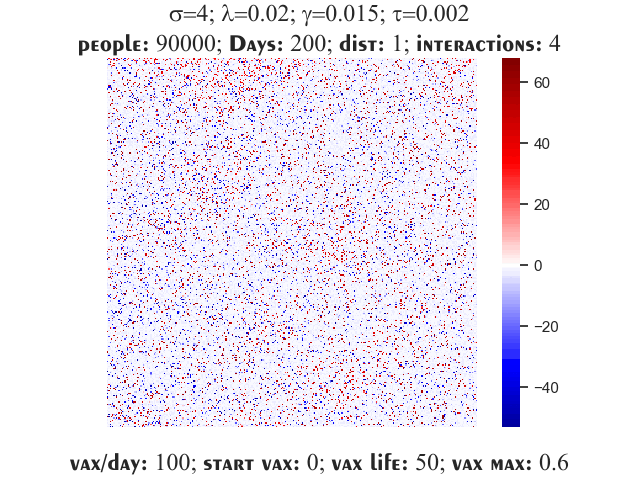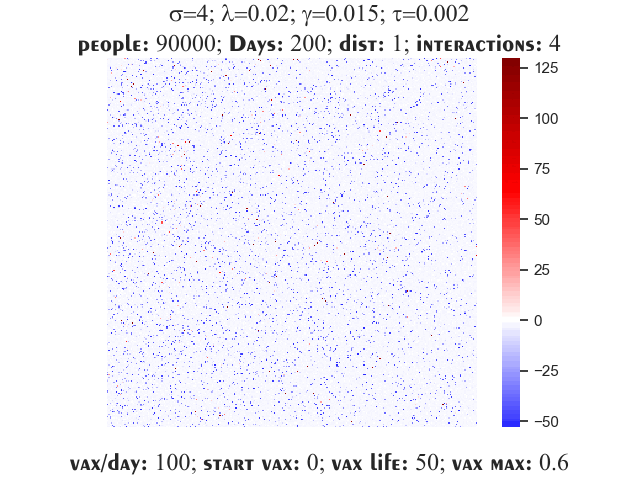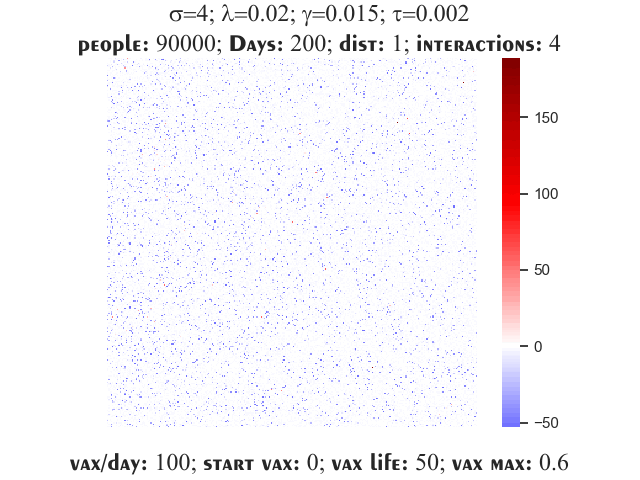
I parametri
Le tre versioni in cui è disponibile ora SIR_ECHovid (Blockly, Python e fogli Google) condividono tutte lo stesso set di parametri:
- Sigma (σ): fattore moltiplicativo del tasso di contagio, per tener conto della diminuzione del tasso dovuta alla distanza, insieme a Distanziamento
- Lambda (λ): tasso di contagio per ogni infettivo nell’unità di tempo
- Gamma (γ): tasso di guarigione (1/γ è la durata media della malattia)
- Tau (τ): tasso di mortalità per ogni infettivo nell’unità di tempo
- Distanziamento: permette di introdurre nel modello interventi di distanziamento sociale simulati (se distanziamento è pari a zero non ci sono limitazioni alle interazioni tra individui)
- Interazioni: controlla il numero di interazioni giornaliere per ogni individuo (in pratica, il numero di persone incontrate, in media, da ogni individuo della popolazione simulata)
Lo stato del singolo individuo, identificato dalla sua posizione riga, colonna nella matrice, è indicato da un numero intero:
- suscettibile: 0
- infettivo: un valore a partire da 1, dove il numero indica i giorni di malattia
- deceduto: -1
- guarito: -2 (sia i deceduti che i guariti rientrano nella categoria rimossi del modello SIR, ma vengono registrati separatamente per una conoscenza più completa del fenomeno e per eventuali sviluppi futuri del progetto che includano vaccini, un’immunità solamente temporanea in caso di guarigione o altre funzionalità aggiuntive)
Prima di lanciare l’app
Per lanciare l’applicazione occorre chiamare preventivamente tre funzioni:
- Inizializzazione: la funzione che assegna i valori ai parametri del modello (infatti ha come parametri proprio quelli elencati qui sopra)
- CreaMatrice: crea la matrice che conterrà la popolazione, il numero di individui sarà pari al prodotto di righe x colonne della matrice (i parametri r -le righe – e c – le colonne della matrice – definiscono infatti le dimensioni della matrice)
- SituazioneInizialeCasuale: crea una situazione iniziale distribuendo tra la popolazione il numero desiderato di infettivi (determinato dal parametro infettivi)
Lanciare l’applicazione
Una volta inizializzati i parametri del modello, creata la popolazione (la matrice) e distribuiti gli infettivi è possibile far partire la simulazione con la funzione SimulazioneSIR i cui parametri sono:
- m: la matrice della popolazione
- durata: la durata, in giorni, della simulazione
- output_parziale: da impostare a vero – True – se si vuole in output la situazione giorno per giorno (se questo parametro è settato a falso – False – l’applicazione fornisce solo i risultati finali)
Output
Terminata la simulazione il numero di infettivi, suscettibili e deceduti è memorizzato in tre liste che hanno un elemento per ogni giorno della simulazione:
- infettivi_storico: il numero di infettivi giorno per giorno
- deceduti_storico: il numero di deceduti in ciascun giorno della simulazione
- guariti_storico: come sopra ma per i guariti
Inoltre è possibile visualizzare lo stato di tutta la popolazione attraverso le funzioni StampaMatrice e TestoMatrice.
Le matrici
SIR_ECHovid utilizza le funzioni per la gestione delle matrici realizzate sempre nell’ambito del progetto RiA e descritte nell’articolo Blockly: le matrici.
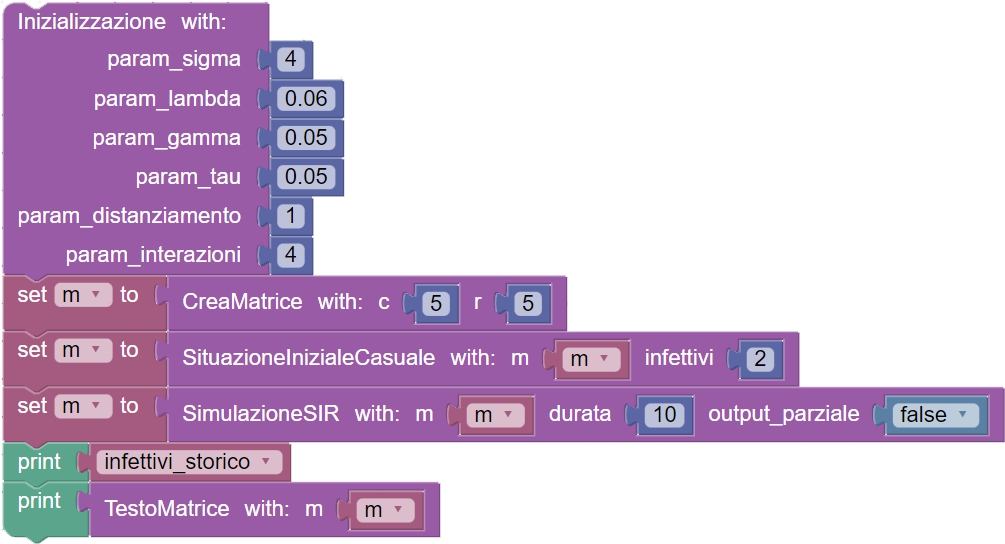
Python
In Python lo stesso lancio ha l’aspetto che segue:
Inizializzazione(4, 0.06, 0.05, 0.05, 1, 4)
m = CreaMatrice(5, 5)
m = SituazioneInizialeCasuale(m, 2)
m = SimulazioneSIR(m, 10, False)
print(infettivi_storico)
print(TestoMatrice(m))Anche in questo caso si chiede all’applicazione di stampare la lista degli infettivi di ogni giorno e la situazione finale della popolazione.
Il foglio di calcolo
Il codice JS generato da Blockly è stato immerso in un foglio di calcolo Google. Per ovvi motivi non è consentita la modifica del foglio di calcolo ma è sufficiente farne una copia sul proprio Drive per accedere a tutte le funzionalità.
Il foglio di calcolo ora ha tre nuove funzioni che permettono di utilizzare il modello agent-based sulla falsariga dei principali modelli teorici SI, SIS e SIR. Per ciascuna delle funzioni i parametri hanno il significato indicato all’inizio dell’articolo:
- ModelloSI(sigma; lambda; distanziamento; interazioni; durata; righe; colonne; infettivi iniziali): simula un’epidemia secondo il modello SI (Suscettibili – Infettivi)
- ModelloSIS(sigma; lambda; distanziamento; interazioni; durata; righe; colonne; infettivi iniziali, gamma): simula un’epidemia secondo il modello SIS (Suscettibili – Infettivi – Suscettibili), ha gli stessi parametri del precedente ma aggiunte il tasso di guarigione gamma
- ModelloSIR(sigma; lambda; distanziamento; interazioni; durata; righe; colonne; infettivi iniziali, gamma, tau): simula il modello SIR (Suscettibili – Infettivi – Rimossi), il parametro ulteriore è il tasso di mortalità tau.
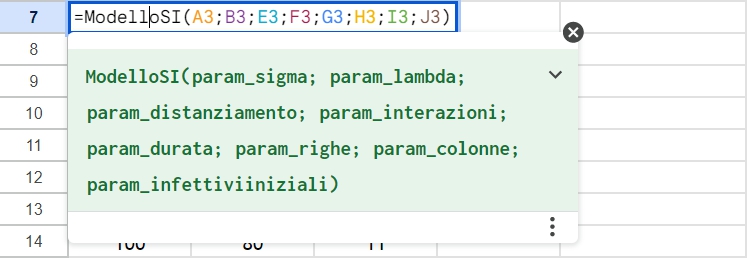
L’articolo Programmare senza un linguaggio di programmazione 3/3 illustra, in breve, come utilizzare il codice JScript generato da Blockly per aggiungere funzionalità a un foglio di calcolo Google.
.